Protein-Design: Biomoleküle optimieren und entwerfen
Proteine sind große biologische Moleküle mit einem hohen Molekulargewicht. Die komplexen Eiweißmoleküle werden gerne auch als molekulare Maschinen bezeichnet, die Lebensprozesse in Betrieb halten. In Zellen und Organismen übernehmen sie vielfältige Aufgaben: Sie bilden unter anderem Zellstrukturen aus (zum Beispiel das Cytoskelett), wirken als Beschleuniger von biochemischen Stoffwechselreaktionen (Enzyme), transportieren Substanzen (Transportproteine) oder erkennen Reize und leiten sie weiter (Rezeptoren und Sensoren). Auch als Energiespeicher, im Immunsystem und der Genregulation übernehmen Proteine zentrale Aufgaben.
Proteine spielen in zahlreichen Anwendungen in Wissenschaft und Wirtschaft eine bedeutende Rolle: So sind Enzyme bedeutende Spezialwerkzeuge in der Biotechnologie. Ihre enorme Anwendungsvielfalt wird in einem anderen Themendossier näher beleuchtet. Biotechnisch erzeugte Proteine werden zudem als Medikamente eingesetzt (etwa Antikörper oder das Peptidhormon Insulin) und sind für zahlreiche biochemische Testverfahren bedeutsam.
Biotechnologen setzen dabei nicht mehr nur auf natürlich vorkommende Proteine. Mithilfe neuester Erkenntnisse und Technologien können sie existierende Proteine in ihrer Funktion verbessern und für einen Anwendungsbereich maßschneidern. Beim Protein Engineering werden Proteine gezielt für eine Aufgabe optimiert oder neu konstruiert (im Folgenden am Beispiel von Enzymen erläutert).
Es gibt zwei unterschiedliche Ansätze:
- Das rationale Protein-Design: Aufbauend auf Daten zur dreidimensionalen Struktur eines Enzyms, die etwa mittels Röntgenkristallographie gewonnen wurden, werden computergestützt Vorhersagen getroffen, an welchen Stellen sich die Eigenschaften des Enzyms optimieren lassen. Nach der Modellierung wird der molekulare Bauplan des Enzyms dann im Labor gezielt verändert und die auf dieser Basis erzeugten Enzym-Varianten werden experimentell überprüft.
- Die gerichtete Evolution: Für diesen Ansatz werden keine dreidimensionalen Strukturdaten der Enzyme benötigt. Vielmehr wird ein Evolutionsprozess im Labor experimentell nachgestellt: Im molekularen Bauplan, dem entsprechenden Gen des Enzyms, werden zufällige Mutationen ausgelöst. Dadurch entsteht eine große Zahl an Enzym-Varianten. Sie werden im Labor dann in Aktivitäts-Testreihen auf verbesserte Eigenschaften geprüft (Selektionsschritt). Die besten Kandidaten durchlaufen weitere Optimierungsrunden.
Innerhalb weniger Monate lassen sich heute mit diesen Ansätzen Enzyme an neue Substrate oder gewünschte Prozessbedingungen anpassen. Kombiniert mit Schlüsseltechnologien wie Künstliche Intelligenz (KI) sowie effizienten Hochdurchsatzverfahren können die Entwicklungszeiten noch weiter verkürzt werden.
Dieses Dossier beleuchtet, wie KI-Technologien die Strukturbiologie und das Protein-Design in den vergangenen Jahren grundlegend verändert haben.
Seite 2 von 6
KI als Schlüsseltechnologie
Künstliche Intelligenz (KI) ist eine Schlüsseltechnologie des 21. Jahrhunderts, die weltweit Wirtschaften und Gesellschaften transformiert. KI birgt dabei als breit einsetzbares Werkzeug vielfältige Chancen. Sie ist dabei, den Umgang mit Informationen und Wissen in Gesellschaft, Wissenschaft und Wirtschaft wesentlich zu verändern. Öffentlichkeitswirksame Erfolge von KI beruhen vor allem auf Methoden des maschinellen Lernens in Verbindung mit einem deutlichen Wachstum der verfügbaren und nutzbaren Daten sowie einer großen und stetig wachsenden Rechenleistung.
Seitdem das US-amerikanische Unternehmen OpenAI im November 2022 den Zugang zu seinem Sprachmodell ChatGPT öffnete, ist KI erstmalig von Millionen von Nutzerinnen und Nutzern getestet und genutzt worden. Spätestens seit diesem Meilenstein der KI-Entwicklung ist generative KI zum Technologiethema Nr. 1 avanciert.
Kurzes KI-Glossar
Künstliche Intelligenz (KI) als Begriff steht für Software- und Robotiksysteme, die ein Verhalten zeigen, für das gemeinhin menschliche Intelligenz vorausgesetzt wird. Zugleich beschreibt KI ein Teilgebiet der Informatik, das sich mit der Entwicklung dieser Systeme beschäftigt.
Maschinelles Lernen ist ein Teilbereich der KI. Dabei lernen Computer durch ein entsprechendes Training, Muster und Gesetzmäßigkeiten in einer großen Datenmenge quasi zu „erkennen“.
Generative KI ist ein Modell des maschinellen Lernens, das anhand von Beispielen (Trainingsdaten) lernt und dann Inhalte generieren kann.
Große Sprachmodelle (engl. Large Language Models, LLMs) sind generative KI-Modelle, die mit riesigen Textmengen trainiert werden. Sie sind in der Lage, „natürlich klingende“ Sprache zu erzeugen, indem sie voraussagen können, welches Wort am wahrscheinlichsten als nächstes kommt.
(Quelle: Plattform Lernende Systeme und Google Aufbruch Nr. 30)
Die Bundesregierung hat im Jahr 2018 eine KI-Strategie veröffentlicht, die mit dem KI-Aktionsplan des BMBF im Jahr 2023 ein Update erhalten hat. Allein das BMBF investiert in KI mehr als 1,6 Mrd. Euro in der laufenden Legislaturperiode mit den Schwerpunkten Forschung, Kompetenzentwicklung, Aufbau von Infrastrukturen und Transfer in die Anwendung.
Auch die im Februar 2023 vom Bundeskabinett beschlossene Zukunftsstrategie Forschung und Innovation enthält eine Reihe von KI-Bezügen. So sollen Transformationsprozesse mithilfe von KI aktiv gestaltet und die technologische Souveränität Deutschlands gewahrt werden. Wie KI in für die Bioökonomie relevanten Bereichen eingesetzt wird, erläutert auch das Themendossier: Bioökonomie und Künstliche Intelligenz.
Seite 3 von 6
Die räumliche Gestalt von Proteinen ermitteln
Proteine sind lange Molekülketten, die aus Aminosäuren als Bausteine bestehen. In aller Regel liegen die Aminosäureketten aber nicht fadenförmig vor, sondern sie verdrehen und falten sich zu komplexen dreidimensionalen Gebilden.
Welche Gestalt sie dabei annehmen, hängt von der Art und Reihenfolge ihrer Aminosäuren ab – denn diese besitzen unterschiedliche Anziehungs- oder Abstoßungskräfte. Die Wechselwirkungen zwischen den diversen Aminosäuren eines Proteins sind meist derart komplex, dass das Verständnis der zugrundeliegenden Prinzipien jahrzehntelange Forschung benötigte.
Anhand der DNA-Sequenz eines Gens lässt sich die Reihenfolge der Aminosäure-Bausteine eines Proteins herleiten – die sogenannte Primärstruktur. Welche biologische Funktion ein Protein hat, hängt jedoch entscheidend von der Faltung (Sekundärstruktur) und der resultierenden räumlichen Gestalt ab (Tertiärstruktur) und der Art der Interaktion mit anderen Proteinen (Quartärstruktur) ab. Die räumliche Struktur ist es, die über die Möglichkeiten eines Proteins zur Wechselwirkung mit anderen Proteinen oder Zellbestandteilen entscheidet.
Experimentelle Strukturbiologie: Datenfutter für die Algorithmen
Die Struktur eines Proteins experimentell aufzuklären, ist sehr aufwendig. Im Prinzip geht es dabei darum, die einzelnen Atome und ihre Position im Raum zu ermitteln. Dazu muss das hochdynamische Protein – beispielsweise ein Enzym – jedoch erst einmal fixiert werden. Vor allem drei Verfahren liefern riesige, hochkomplexe Datensätzen – die nur computergestützt ausgewertet werden können.
Die Röntgenkristallographie untersucht Moleküle, die zuvor kristallisieren müssen. Anhand der Beugung der Strahlung lässt sich dann die Struktur des Kristalls errechnen. Heute ist das Verfahren eine Standardmethode in der Strukturbiologie und für rund 90 % der experimentell bestimmten Proteinstrukturen genutzt worden.
Bei der Kryo-Elektronenmikroskopie wird ein Enzym ultraschnell bei –150 Grad Celsius eingefroren, sodass es nicht mehr in Bewegung ist. Dann wird eine elektronenmikroskopische Aufnahme erzeugt.
Die Kernspinresonanz-Spektroskopie ist vor allem deshalb interessant, weil sie es ermöglicht, Enzyme in ihrer Dynamik und möglichen unterschiedlichen Konformationszuständen zu erfassen.
Seite 4 von 6
Proteinstrukturen mit smarter Software vorhersagen
Generell benötigen die experimentellen Verfahren der Strukturaufklärung von Proteinen teure Geräte, viel Zeit und sind trotzdem mit Unsicherheiten behaftet. Forschende in der Strukturbiologie suchten schon seit Langem nach Wegen, die Molekülstruktur theoretisch aus der Sequenz der einzelnen Aminosäure-Bausteine herzuleiten.
Doch ein Protein kann aus hunderten bis tausenden Aminosäuren bestehen. Die astronomisch hohe Zahl möglicher Kombinationen von Aminosäuren und ihrer Wechselwirkungen untereinander macht die Erforschung der 3D-Struktur zu einem gewaltigen Puzzlespiel. Der Vorgang ist selbst mit moderner Computertechnik sehr zeitaufwendig. Um jedoch anhand der Primärstruktur eines Proteins zu errechnen, wie sich das Protein falten wird, sind enorme Rechenkapazitäten erforderlich. Die Idee dabei ist, dass die Gesetze der Chemie nur bestimmte Optionen zulassen und ein Protein aus diesen Optionen wohl die Faltung einnehmen wird, die energetisch die günstigste ist.
Proteinfaltung mit vereinter Rechenpower
Im Jahr 2000 startete dazu das Projekt Folding@Home, entwickelt von einem Team der Standford University. Hier werden über das Internet Tausende private Computer vernetzt, die immer, wenn sie im Leerlauf sind, ihre Ressourcen für das Forschungswerkzeug bereitstellen. Folding@Home sucht als Citizen-Science-Projekt vor allem nach den Ursachen für Protein-Fehlfaltungen, die mit Erkrankungen wie Alzheimer oder Huntington zusammenhängen. Die Grundlage dafür bilden sogenannte Markov-Modelle, die die Wahrscheinlichkeitstheorie verwenden, um sich zufällig verändernde Systeme zu modellieren.
Auf ein ähnliches Prinzip wie Folding@Home setzt Rosetta@Home, das 2005 von dem Team um David Baker an der University of Washington in Seattle gestartet wurde. Es wurde auf der Basis der Software BOINC entwickelt. Rosetta unterteilt ein Protein in kurze Abschnitte von Aminosäureabfolgen, für die es ähnliche Sequenzen bei anderen Proteinen gibt, von denen die entsprechende Raumstruktur bekannt ist. Aus den so ermittelten Einzelstrukturen baut die Software nach und nach die Gesamtstruktur zusammen. Das Baker Lab verfolgt mit Rosetta das Ziel, nicht nur häufig zu einem zutreffenden Ergebnis zu gelangen, sondern exakte Strukturvorhersagen zu machen. Nur dann lässt sich das Werkzeug nutzen, um neue Proteine zu designen.
Rund 44.000 Computer stellen derzeit Rosetta ihre freie Rechenleistung zur Verfügung, und ähnlich wie bei Folding@Home sorgte die COVID-19-Pandemie für einen Leistungsschub. Bis 2020 galt Rosetta als der Goldstandard, um Proteinstrukturen vorherzusagen. Trotzdem kam die Software bei der Genauigkeit ihrer Vorhersagen den experimentell ermittelten Strukturen nur mäßig nah.
KI-Revolution in der Proteinfaltungsforschung
Künstliche Intelligenz mit ihrer besonderen Stärke in der Mustererkennung hat in der Proteinfaltungsforschung eine neue Ära eingeläutet. Machine-Learning-Algorithmen können immer besser vorhersagen, wie sich ein Protein aufgrund seiner Aminosäure-Sequenz dreidimensional zu einem Knäuel falten wird.
Bahnbrechend für die Proteinforschung war 2020 die Veröffentlichung von AlphaFold, einem Deep-Learning-Algorithmus des britischen Unternehmens Deepmind. Die KI wurde mit allen bis dahin experimentell ermittelten Proteindaten der Protein Data Bank (PDB) trainiert: Dazu fütterten die Entwickler den Algorithmus mit der Aminosäuresequenz von 170.000 Proteinen und deren zugehörigen Raumstruktur. Daraus hat die Software Muster und Zusammenhänge abgeleitet, die sie nutzt, um für unbekannte Sequenzen die 3D-Struktur vorherzusagen.
Wie leistungsfähig die Software ist, stellte sie in dem alle zwei Jahre stattfindenden Wissenschaftswettbewerb für computergestützte Strukturbiologie CASP (Critical Assessment of Structure Prediction) eindrucksvoll unter Beweis. Das Ziel bei CASP: Bioinformatiker rund um den Globus reichen hier ihre computergestützten Strukturvorhersagen für die räumliche Gestalt biologischer Moleküle ein, für die zwar bereits experimentelle Strukturdaten vorliegen, aber noch nicht veröffentlicht sind.
AlphaFold im Jahr 2018 und insbesondere der Nachfolger AlphaFold2 im Jahr 2020 ließen bei CASP alle weiteren mitwirkenden Teams weit hinter sich. Sie konnten mit hoher Verlässlichkeit vorherzusagen, wie sich eine Aminosäure-Sequenz falten würde. Das Fachjournal „Science“ kürte Alphafold zum wissenschaftlichen Durchbruch des Jahres 2021. Inzwischen gehört Deepmind zum Alphabet-Konzern, der AlphaFold2 als Open-Source-Lizenz der Allgemeinheit zur Verfügung gestellt hat.
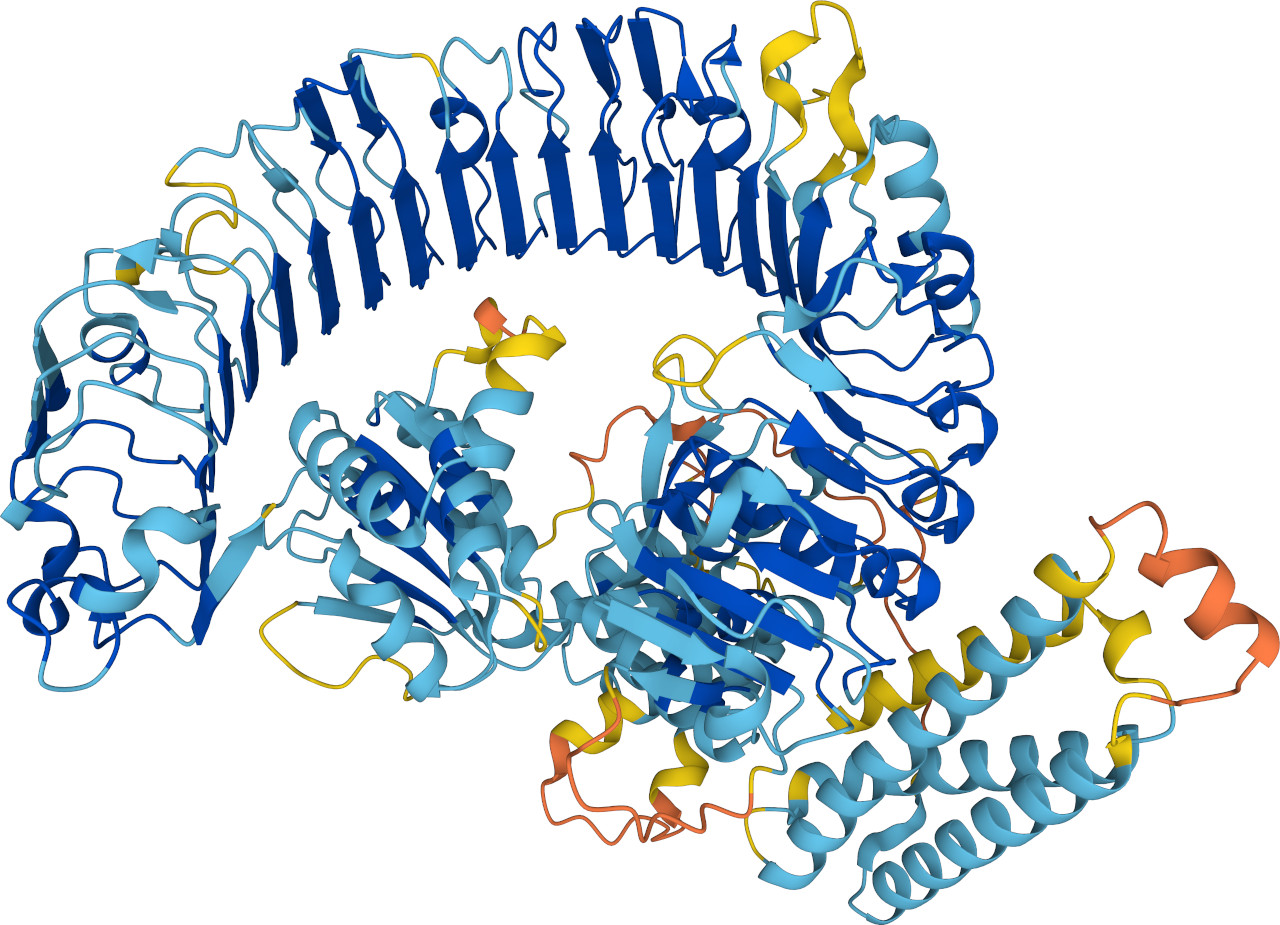
200 Millionen Proteinstrukturen simuliert
Gemeinsam mit dem Europäischen Institut für Bioinformatik (EMBL-EBI) hat das DeepMind-Team die AlphaFold Proteinstrukturdatenbank aufgebaut. Unter anderen wurde AlphaFold2 genutzt, um die Struktur der Proteine des COVID-19-Erregers SARS-CoV-2 zu ermitteln. So konnte man Impfstoffe und Medikamente herstellen. Seit dem 28. Juli 2022 umfasst die Datenbank die Strukturvorhersagen für rund 200 Millionen Proteinmodelle.
Das Baker Lab hat im Jahr 2021 nachgezogen und mit RoseTTAFold eine Software vorgestellt, die eine ähnliche Präzision erreicht wie AlphaFold2.
Dennoch hat AlphaFold2 einige Schwächen. So konnte sie lange Zeit nur Monomere vorhersagen, nicht jedoch Proteinkomplexe. Ebenso wenig berücksichtigt der Algorithmus Cofaktoren. Das soll jetzt ein separates Modell namens AlphaFill leisten. Außerdem beruht das Modell teilweise darauf, dass ähnliche Proteine eine ähnliche Evolution verbindet. In Bezug auf Vorhersagen für synthetische Proteine, für die diese Co-Evolution nicht existiert, könnte das zu Fehlern führen. Nicht zuletzt ermittelt AlphaFold2 immer nur eine Struktur für ein Protein, obwohl Proteine durchaus in mehreren Faltungen existieren können.
Während AlphaFold2 beispielsweise eine Vorstellung von der Struktur eines Proteins vermittelt, liefern Röntgenstrahlen und hochauflösende Kryo-Elektronenmikroskopie in der Regel immer noch die genaueren Strukturen, die für das Verständnis enzymatischer Reaktionen oder die Entwicklung von Medikamenten benötigt werden. Dennoch sind AlphaFold2 und RoseTTaFold innerhalb kürzester Zeit zum integralen Bestandteil der Arbeit von Strukturbiologen und Enzymdesignern weltweit geworden und haben die Proteinforschung transformiert.
Stärken und Schwächen von AlphaFold2 im Überblick
Was AlphaFold2 vorhersagen kann: Faltung einzelner Proteinketten, Protein-Multimere, Protein-Protein-Komplexe mit mehreren Untereinheiten
Hier hat AlphaFold2 Schwierigkeiten bei den Vorhersagen: verschiedene Konformationen der gleichen Aminosäuresequenz; Auswirkungen von Punktmutationen; Antigen-Antikörper-Wechselwirkungen
Was AlphaFold nicht vorhersagen kann: Protein-DNA sowie Protein-RNA-Komplexe, Nukleinsäure-Strukturen, Liganden-Ionenbindung, posttranslationale Modifikationen; die angrenzende Membranschicht bei Transmembran-Domänen der Proteine
Quelle: EMBL-EBI Training
Seite 5 von 6
Proteinstrukturvorhersagen mit großen Sprachmodellen
Die Veröffentlichung von AlphaFold2 im Juli 2021 war für Strukturbiologie bahnbrechend. Im November 2022 folgte der „ChatGPT-Moment“: Das Unternehmen OpenAI veröffentlichte das große Sprachmodell GPT und das auf ihm basierenden Chatbot und katapultierte damit das Thema generative KI in die Mitte der Gesellschaft.
GPT steht für Generative Pre-Trained Transformer. Das Modell basiert auf einer neuronalen Netzwerk-Architektur, die der Struktur des menschlichen Gehirns ähnelt, und daher Transformer-Architektur genannt wird.
Salopp sprechen Fachleute bei den großen Sprachmodellen (Large Language Model) von stochastischen Papageien. Denn das Prinzip ist überraschend simpel: GPT-4, die 2023 aktuelle Form, hat – vereinfacht gesagt – ganz viel gelesen. Wikipedia-Einträge, das Gutenberg-Projekt, Bücher, Briefe und mehr. Auf dieser Grundlage ermittelt der Chatbot Buchstabe für Buchstabe, wie die wohl wahrscheinlichste Antwort auf eine gestellte Frage oder Aufgabe aussieht. In der Trainingsphase erhielt das Programm dann Rückmeldung zu seinen Antworten durch Menschen, um dadurch besser zu werden. Weil ChatGPT rein stochastisch Buchstabe um Buchstaben aneinanderreiht, hat es kein Verständnis vom Inhalt seiner Antworten. Wo es keine Antworten findet, „halluziniert“ es erfundene Antworten und Quellen. Trotzdem erreicht ChatGPT eine bis dahin ungekannte Funktionalität.
Sprachmodelle für die Proteinstruktur-Vorhersage
Nach dem gleichen Prinzip, auf dem GPT basiert, hat 2022 der Facebook-Mutterkonzern Meta ESMFold vorgestellt, ein Large-Language-Modell zur Proteinstruktur-Vorhersage. Anstatt ausgehend von den Buchstaben des Alphabets auf stochastische Weise Sätze zu bilden, verwendet ESMFold die Aminosäuren als Buchstaben und setzt daraus Proteine zusammen. Um sich die „Sprache der Proteine“ zu erschließen, lernte das Programm zunächst, „Lückentexte“ zu füllen, also in Aminosäure-Sequenzen Auslassungen korrekt zu befüllen. So entwickelte die KI eine Art intuitives Verständnis von Proteinsequenzen.
In einem zweiten Schritt kombiniert sie dann – ähnlich wie AlphaFold2 – dieses Verständnis mit dem Wissen über die Wechselbeziehungen zwischen Sequenzen und Strukturen, das aus experimentell bestimmten Proteinstrukturen stammt, aber auch aus Strukturen, die AlphaFold2 vorhergesagt hat.
Stand 2023 ist ESMFold etwas weniger präzise als AlphaFold2. Aber insbesondere bei kürzeren Sequenzen mit bis zu 1024 Aminosäuren ist der Algorithmus 60-mal so schnell. Innerhalb von zwei Wochen machte ESMFold Strukturvorhersagen für 617 Millionen Proteine. Untersucht wurden dabei die Daten sogenannter metagenomischer DNA aus Umweltproben – als einem großen Mix des Erbguts unzähliger Mikroorganismen, die größtenteils noch nie kultiviert worden sind. Verfügbar sind die Daten im ESM Metagenomic Atlas, inzwischen summieren sie sich auf 772 Millionen Proteine.
Etwa ein Drittel dieser Vorhersagen gelten als so gut, dass sie oftmals bis ins atomare Detail hinein stimmen. Millionen Strukturen sind jedoch gänzlich anders als das, was bislang aus der Forschung bekannt ist, die Strukturprognosen von AlphaFold2 eingeschlossen. Das deutet darauf hin, dass die Welt der Proteine innerhalb der noch unerforschten Mikroorganismen weit vielfältiger ist als das, was bislang in den Laboren der Welt untersucht werden konnte.
In eine ähnliche Richtung deuten die vielen Millionen Strukturen, denen ESMFold selbst eine geringe Verlässlichkeit zuordnet: Haben zahlreiche Proteine vielleicht nicht die eine definierte Struktur, sondern sind hochdynamisch? In jedem Fall gehen viele Fachleute davon aus, dass ein solches KI-Modell besonders gut geeignet ist, um vorherzusagen, wie sich ein Protein verändert, wenn man einzelne Aminosäuren austauscht.
Nicht erfüllt hat sich bislang die Erwartung, dass Sprachmodelle Proteine, zu denen keine verwandten Moleküle bekannt sind, besser vorhersagen können als AlphaFold2 und RoseTTAFold. Schließlich beruhen letztere stark auf Sequenz-Analogien. Beim jüngsten CASP-Wettbewerb zeigte sich ESMFold diesbezüglich nicht überlegen.
Seite 6 von 6
Mit KI-Modellen neuartige Designer-Proteine entwerfen
Proteinstrukturen zu verstehen und Enzyme so zu optimieren, dass sie spezifischer oder mit höheren Umsatzraten arbeiten, ist in der Medizin ebenso wichtig wie in der industriellen Biotechnologie. Unter Synthetischer Biologie versteht man Ansätze zur gezielten Erzeugung biologischer Systeme mit neuen Eigenschaften (mehr in diesem Themendossier). Dazu zählt auch, künstliche Proteine herzustellen, die speziell für einen Zweck optimal designt sind. Lange Zeit spielten dabei Versuch und Irrtum eine wichtige Rolle, wenn Forschende Strukturen von Abschnitten bekannter Proteine kombinierten. Inzwischen ist auch dieses Forschungsfeld von KI-Technologien dominiert.
Sprachmodelle für das Protein-Design
Seit 2022 sind zahlreiche Protein-Sprachmodelle entstanden. Für zahlreiche Proteinklassen können inzwischen mit der gleichen Genauigkeit Strukturen am Computer ermittelt oder generiert werden wie durch experimentelle Verfahren. Schwierigkeiten bereiten vor allem noch „intrinsisch ungeordnete Proteine“, also flexible Moleküle ohne feste Struktur, Proteine mit Cofaktoren und große Protein-Komplexe. Chancen hingegen sehen Fachleute etwa bei neuartigen Sequenzen, die weit von bekannten Strukturen abweichen und trotzdem funktional sein könnten.
Das David Baker Lab etwa brachte RFdiffusion heraus, um Proteine zu erschaffen, wie sie die Evolution nicht hervorgebracht hat. Obwohl die Software auf einem Protein-Sprachmodell basiert und damit stochastisch arbeitet, scheint sie viele Regeln der Proteinfaltung besser zu beherrschen als ihre Schöpfer.
Ein Team um Burkhard Rost von der TU München hat das Protein-Sprachmodell EMBER2 entwickelt. Auch hier legten die Forscher den Fokus darauf, dass die Software unabhängig von Sequenz-Analogien funktioniert. Dadurch erreicht das Modell nicht ganz die Qualität von AlphaFold2. Aber EMBER2 kann ungewöhnliche Proteinstruktur besonders gut vorhersagen und eben auch neuartige Proteine designen – zu geringeren Kosten als die Konkurrenz aus dem Hause Alphabet.
Inzwischen gibt es zahlreiche Sprachmodelle, die auch für das Protein-Design genutzt werden – darunter sind ProGen, Chroma oder ProtGPT2.
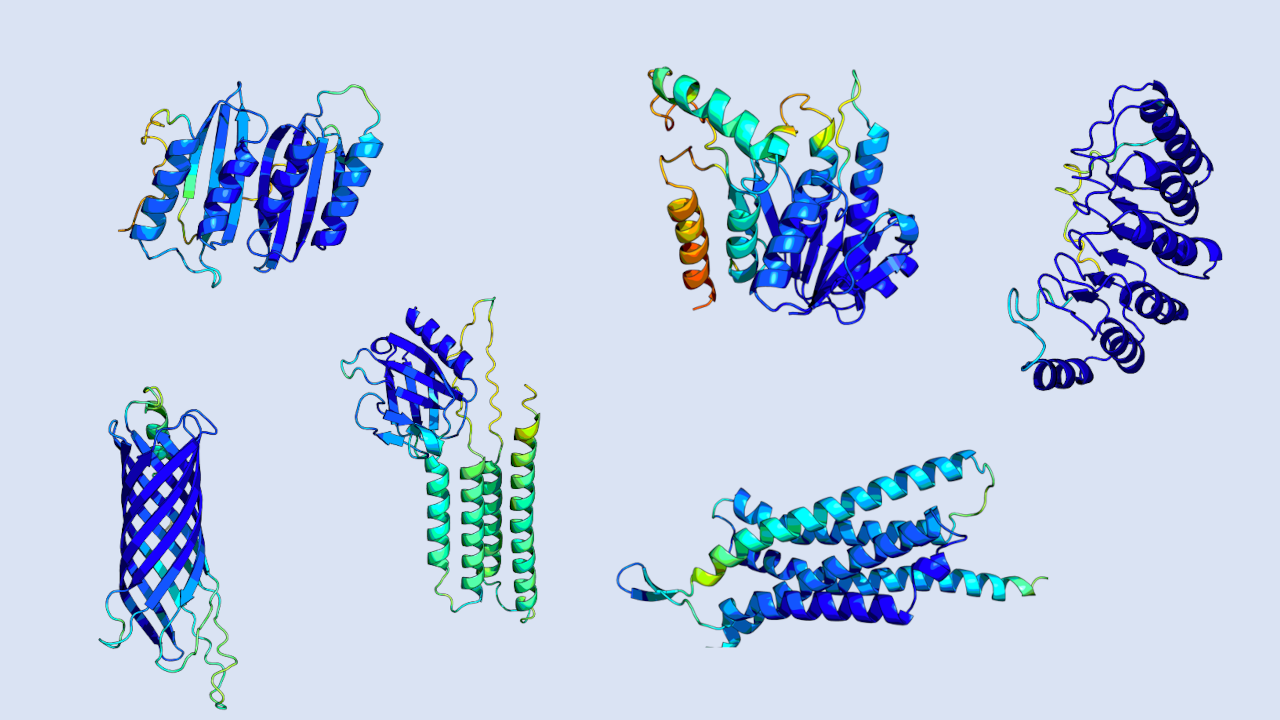
Mit ProtGPT2 Proteine kreieren
ProtGPT2 wurde von dem Team um Birte Höcker von der Universität Bayreuth entwickelt. Das Sprachverarbeitungsmodell hat ihr Team mit 50 Millionen Sequenzen natürlicher Proteine trainiert. „Nun versteht es nicht nur die Sprache der Proteine, sondern kann sie auch kreativ anwenden“, sagt Birte Höcker im Interview mit bioökonomie.de.
Jetzt ließen sich damit Proteine entwerfen, die durch Faltung stabile Strukturen annehmen und in diesem Zustand dauerhaft funktionstüchtig sind, sagt Höcker. Als Besonderheit erzeuge ProtGPT2 Proteine, die von Hause aus eine derart ausdifferenzierte Struktur besitzen, dass sie in ihrer jeweiligen Umgebung bereits einsatzfähig sind. „Wir haben zudem Hinweise, dass das Modell Proteine kreieren kann, die in der Natur nicht vorkommen und in der Geschichte der Evolution womöglich noch nie existiert haben“, so Höcker. „Das öffnet die Tür zu einer innovativen Forschung, die bisher unbekannte Proteine auf neuartige Weise erzeugt.“
Nicht alle KI-Werkzeuge allgemein zugänglich
Sorge bereitet insbesondere akademischen Forscherinnen und Forschern, aber auch Start-ups, dass sieben der neun heute für Proteine genutzten Transformer von Großkonzernen entwickelt wurden. Das liegt vor allem an den enormen Kosten und der benötigten Rechenleistung, die das Training der KI erfordert. Immerhin habe manche Unternehmen ihr Produkt frei verfügbar gemacht, wie AlphaFold2 und ProtTrans.
So vielversprechend die bisherigen Erfolge der Sprachmodelle sind, neue Proteine zu designen: Es gibt noch viel zu optimieren. Die sogenannten Aufmerksamkeitsmodelle, die den meisten Sprachmodellen ermöglichen, sich auf relevante Teile der Eingabedaten zu fokussieren und diese gezielt für die Verarbeitung zu nutzen, sind nicht für dreidimensionale Strukturen ausgelegt. Die Mehrzahl der Modelle arbeitet am verlässlichsten innerhalb konventioneller Proteinstrukturen und limitiert so das Innovationspotenzial.
Und nicht zuletzt haben große KI-Modelle einen hohen Energiebedarf und damit einen recht hohen CO₂-Fußabdruck. Forschende arbeiten an datensparsameren und energieeffizienteren Modellen.
Neue Wege in der Protein-Forschung eröffnet
Doch auch wenn noch einige Herausforderungen zu bewältigen sind: In der Forschungsgemeinschaft herrscht weitgehend Einigkeit darüber, dass die Arbeit an Proteinstrukturen nicht mehr ohne KI zu denken ist. Zudem seien beim Protein-Design durch technologische Fortschritte die Erfolgsquoten enorm gestiegen, sagt Birte Höcker. „Früher sind viele Designs bereits an der Herstellung – also etwa an der Protein-Expression in Bakterienzellen – gescheitert. Heute beobachten wir eine deutliche Verbesserung in den Eigenschaften und der Handhabung vieler Designer-Proteine, sodass wir ganz neue Fragen stellen und Herausforderungen angehen können.“



